https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3?report=fasta
#2019ncov #chiny #2019ncov #wirus #genetyka #bioinformatyka
Wszystko
Najnowsze
Archiwum
źródło: comment_63OcHa6cMImMkED4pSJrgh2RUkPRxZDh.jpg
Pobierz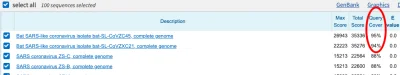
źródło: comment_seueNV2kC9Hht8LHLD5ruPzHzW05eRnf.jpg
Pobierz
źródło: comment_EBW45Sblqwv4B9p8e4pd3MozPEQusOhb.jpg
PobierzKomentarz usunięty przez autora
Komentarz usunięty przez autora Wpisu
Komentarz usunięty przez autora
260
Już 1 mln razy badacze i osoby z całego świata wykorzystały RNAComposer – publicznie dostępny, skuteczny poznański system do modelowania struktury 3D RNA. A to nie jedyny polski sukces w badaniach nad wyznaczaniem struktury RNA.
zKomentarz usunięty przez autora
2

Karin Strauss z Microsoft Research w Redmond uważa swój komputer za osobistą cyfrową bibliotekę, zawierającą wszystkie jej prace jakie napisała począwszy od liceum przez coledge aż do dzisiaj. Chciałaby, aby te dane istaniały jak najdłużej, ale komputery trzeba co parę lat wymieniać a to...
z2
Niemożność dokonania stuprocentowo wiernej symulacji zachowań odpowiednio złożonego systemu (lub inaczej: niemożność zbudowania wiernego w stu procentach modelu pewnej klasy systemów).Dowodem na to ...
z
źródło: comment_u3ya5GUq229FYiHtdt9svTjCnOGYi9Ul.jpg
Pobierzzbior.size() a nie widzę problemów byś miał jakieś rożne rozmiary, zamiast:int ZnajdzMaxWartosc(int c, vector tab)int ZnajdzMaxWartosc(vector const&Regulamin
Reklama
Kontakt
O nas
FAQ
Osiągnięcia
Ranking
#programowanie #bioinformatyka
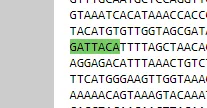
wchodzisz na ncbi.nlm.nih.gov/blast
wybierasz nucletotide blast
odhaczas " Align two or more sequences"
Wklejasz sekwencję pierwszą do pierwszego okienka, drugą do drugiego
i przyrównujesz, najlepiej na domyślnych ustawieniach, bo po co kombinować bardziej