Aktywne Wpisy

Kalosz667 +49
#nieruchomosci #demografia
Wykopki: dziś 300k to gówno nie pieniądz, bez 800k a najlepiej miliona nawet nie myśl o własnym mieszkaniu, demografia przez to upadnie
Tymczasem każde miasto 20k-100k mieszkańców w Polsce:
Wykopki: dziś 300k to gówno nie pieniądz, bez 800k a najlepiej miliona nawet nie myśl o własnym mieszkaniu, demografia przez to upadnie
Tymczasem każde miasto 20k-100k mieszkańców w Polsce:
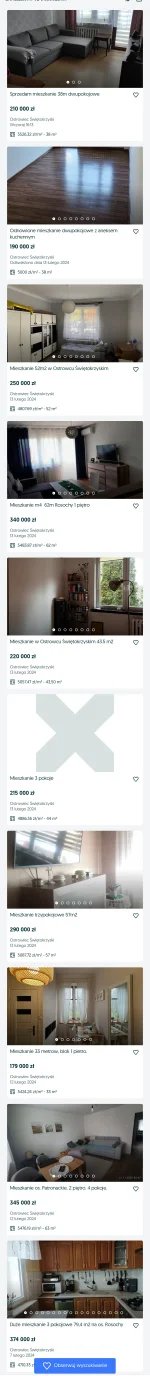
źródło: Screenshot_20240215_122442
Pobierz
WielkiNos +402
Ze wszystkich zdjęć, które wrzucała kiedykolwiek Lewandowska i o których dyskutuje się na wykopie i twitterze to jest dla mnie najgorsze.
Lewandowska na pierwszym planie z sexi dzióbkiem, za nią stado opalonych amigos uśmiechających się sugestywnie, na samym końcu za nimi wszystkimi, w cieniu stoi jeden z największych światowych talentów piłkarskich - Robert Lewandowski.
O ile lepiej by to zdjęcie wyglądało gdyby Lewandowska stanęła obok Roberta, objęła go i pochyliła się w
Lewandowska na pierwszym planie z sexi dzióbkiem, za nią stado opalonych amigos uśmiechających się sugestywnie, na samym końcu za nimi wszystkimi, w cieniu stoi jeden z największych światowych talentów piłkarskich - Robert Lewandowski.
O ile lepiej by to zdjęcie wyglądało gdyby Lewandowska stanęła obok Roberta, objęła go i pochyliła się w

źródło: temp_file8351065228825838349
Pobierz




Uczę się do kolokwium z Bioinformatyki i mam mała zagwozdkę. Treść zadania:
"W oparciu o podaną tabelę kodu genetycznego zmodyfikuj daną sekwencje DNA każdorazowo wprowadzając zmian tak, aby zawierała mutację jednonukleotydową a) cichą/ b) synonimiczną / c) niesynonimiczną / d) nonsens. Opisz, na czym polega ten typ mutacji. Przyjmij założenie, że właściwa ramka odczytu to +1 i przedstawia ona pełną sekwencję białka.
Sekwencja wyjściowa:
ATGCTTATGAGATCGCGAGACGAA"
Ogólnie wiem o co chodzi w zadaniu, mam podzielić sekwencje na kodony i odpowiednio je zmodyfikować. Mam tylko problem ze zrozumieniem o co chodzi z "Przyjmij założenie, że właściwa ramka odczytu to +1 i przedstawia ona pełną sekwencję białka." Ktoś jest w stanie mi to sensownie wytłumaczyć?
(mój kierunek nie ma w ogóle nic wspólnego z bioinformatyką, nie wiem czemu mam ten przedmiot ale kulam tą kupę na zaliczenie. Wiem, że dla kogoś kto w tym siedzi musi to być banalne więc proszę o pomoc.)
1. P.G.Higgs, T.K.Atwood. Bioinformatyka I ewolucja
molekularna. PWN. Warszawa. 2012.
2. J.Xiong. Podstawy bioinformatyki. Wyd.
Uniwersytetu Warszawskiego. Warszawa. 2009.
z tymi ramkami odczytu w sumie to wiem o co chodzi tylko nie byłem pewien na 100% że to o to chodziło.